- オープンデータの衛星画像ALOS/AVNIR2をjupyterバンド演算と図化をしてみました。
- 下の絵はハードコピーですが実際は移動・拡大縮小が可能です。(実はこれが本質だったりします。webベースならインタラクティブなものを使いましょう = matplotlibやめましょう。)
元データ
前処理
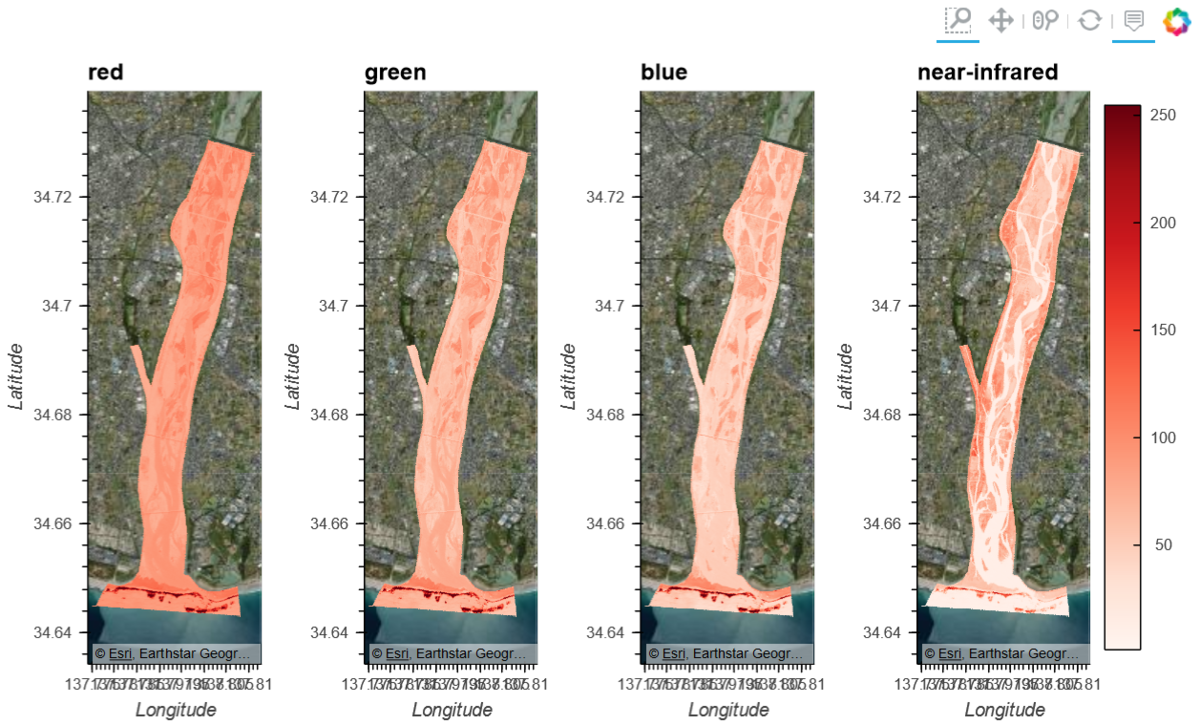
- 元データは4バンド(赤、緑、青、近赤外線)が個別ファイルになっています。
- 前処理は、1).対象領域でクリップ、2).4バンドを1ファイルを行ってます。出力ファイル名:tenryurivmouth.tif
- コードはnbviewerを参照下さい。
- データはgithubを参照下さい。
参考サイト
モジュールを読み込む
import hvplot
import hvplot.xarray
import geoviews as gv
import xarray as xr
import numpy as np
インプットファイルを読み込む
d = xr.open_rasterio('tenryurivmouth.tif')
こんな感じ
geomap = gv.tile_sources.EsriImagery
red = geomap * d.isel(band=0).hvplot.image(x='x',y='y',by='band',cmap='reds',geo=True,frame_height=400,clim=(1,255)).options(title='red',colorbar=False,clipping_colors={'min':'transparent'})
green = geomap * d.isel(band=1).hvplot.image(x='x',y='y',by='band',cmap='reds',geo=True,frame_height=400,clim=(1,255)).options(title='green',colorbar=False,clipping_colors={'min':'transparent'})
blue = geomap * d.isel(band=2).hvplot.image(x='x',y='y',by='band',cmap='reds',geo=True,frame_height=400,clim=(1,255)).options(title='blue',colorbar=False,clipping_colors={'min':'transparent'})
nir = geomap * d.isel(band=3).hvplot.image(x='x',y='y',by='band',cmap='reds',geo=True,frame_height=400,clim=(1,255)).options(title='near-infrared',clipping_colors={'min':'transparent'})
red+green+blue+nir
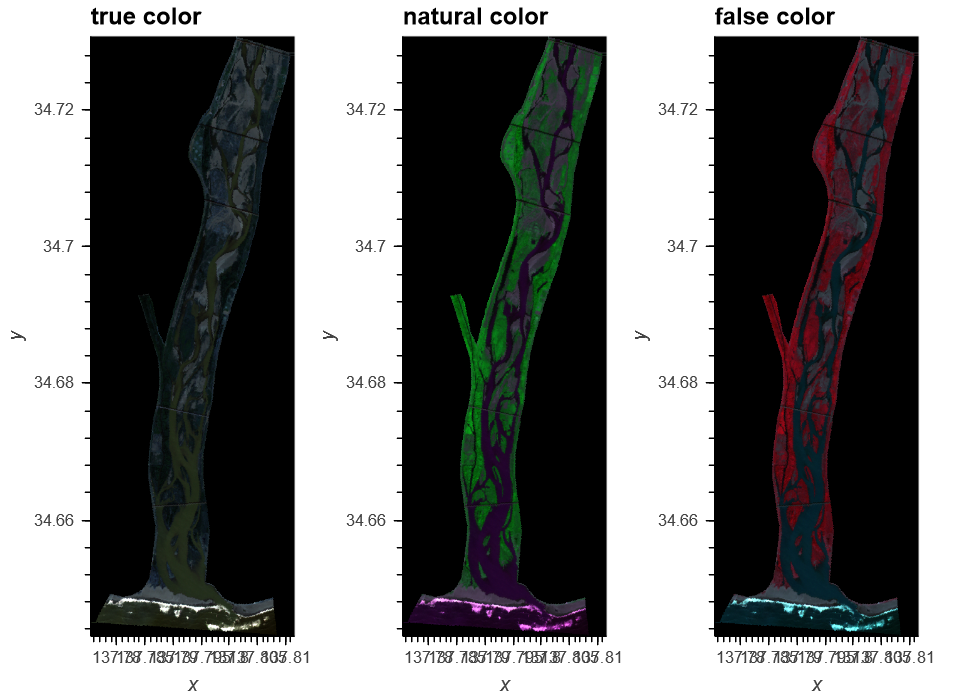
- バンド合成はトゥルーカラー、ナチュラルカラー、フォルスカラーの3パターン。それぞれの定義はwikipediaを参照
- バンド合成では各バンド値を最大値から最小値を引き伸ばすことによる正規化を行っています。
- 元データの型はuintのため解析範囲外は0としてます。正規化処理を行う際に不都合のため、0をNaNに変化するトリッキーな方法を使っています。
dnan = d.where(d>0)
norm = lambda arr : (arr-np.nanmin(arr)) * 255/(np.nanmax(arr)-np.nanmin(arr))
val = np.array([norm(dnan.values[0]), norm(dnan.values[1]), norm(dnan.values[2]), norm(dnan.values[3])])
val = np.where(np.isnan(val), 0, val)
val = val.astype(np.uint8)
dnan.values = val
true = dnan.isel(band=[0,1,2]).hvplot.rgb(x='x',y='y',z='value', bands='band',geo=True, frame_height=400).options(title='true color')
natural = dnan.isel(band=[0,3,1]).hvplot.rgb(x='x',y='y',z='value', bands='band',geo=True, frame_height=400).options(title='natural color')
false = dnan.isel(band=[3,0,1]).hvplot.rgb(x='x',y='y',z='value', bands='band',geo=True, frame_height=400).options(title='false color')
true + natural + false

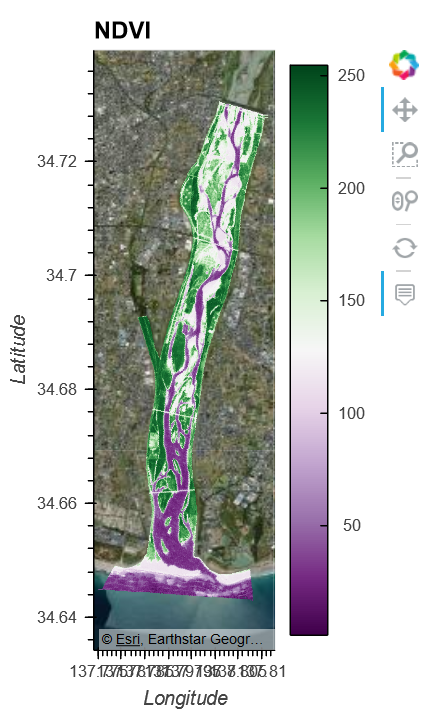
- ラスタ演算はNDVI : Normalized Difference Vegetation Indexのみ。定義はwikipediaを参照
dras= dnan.where(dnan>0)
b = dras.values
NDVI = (b[3] - b[0])/(b[3] + b[0])
NDVI = (NDVI+1)*255/2
NDVI = np.where(np.isnan(NDVI), 0, NDVI)
NDVI = NDVI.astype(np.uint8)
dd = xr.DataArray(NDVI
, coords={'x':d['x'], 'y':d['y']}
, dims=['y', 'x']
, attrs= d.attrs )
geomap = gv.tile_sources.EsriImagery
geomap * dd.hvplot.image(x='x',y='y',cmap='PRGn',geo=True,frame_height=400,clim=(1,255)).options(title='NDVI', clipping_colors={'min':'transparent'})
動作環境
conda create -y -n sate python=3.7
conda activate sate
conda install -y -c conda-forge bokeh=1.4.0
conda install -y -c pyviz geoviews=1.7.0
conda install -y -c pyviz hvplot=0.5.2
conda install -y -c conda-forge rasterio=1.0.21
github.com


